Implementasi klinis dari genomic surveillance berkelanjutan untuk mengidentifikasi, melacak, dan menghentikan transmisi bakteri patogen yang resistan terhadap berbagai obat
Healthcare-associated infections (HAIs) umumnya dikaitkan dengan peningkatan risiko mengembangkan antimicrobial resistance (AMR). Secara global, banyak pasien yang terkena dampak HAIs, yang secara substansial telah meningkatkan keseluruhan biaya operasional sistem perawatan kesehatan. Meskipun sangat penting untuk mengidentifikasi patogen dengan tingkat penularan tinggi di lingkungan rumah sakit, kapasitas laboratorium diagnostik kurang untuk melacaknya.
Latar belakang
Di Australia, lebih dari 165.000 pasien mengalami HAIs setiap tahun. Sebuah survei 30 hari di Australia mengungkapkan bahwa tingkat kematian untuk infeksi methicillin-resistant Staphylococcus aureus (MRSA) dan vancomycin-resistant Enterococcus (VRE) di rumah sakit masing-masing adalah 14,9% dan 20%. Survei yang sama juga melaporkan 18,6% kematian karena infeksi aliran darah extended-spectrum beta-lactamase-producing Escherichia coli di rumah sakit.
Analisis genom telah terbukti menjadi alat yang efektif untuk mengkarakterisasi rute transmisi patogen. Alat ini dapat meningkatkan tindakan pencegahan dan pengendalian infeksi selama wabah patogen. Namun demikian, ini jarang digunakan sebagai alat pengawasan dan pencegahan waktu nyata.
Metode konvensional yang digunakan untuk analisis genetik biasanya memakan waktu dan instrumen analisis tidak mudah tersedia di luar laboratorium khusus. Baru-baru ini, metode whole genome sequencing (WGS) telah dikembangkan untuk menganalisis dinamika transmisi patogen bakteri, yang membantu menilai potensi wabahnya. Metode ini dapat digunakan sebagai alat garis depan untuk mengelola patogen yang dapat mengancam kehidupan manusia.
Dalam studi Clinical Infectious Diseases baru-baru ini, para ilmuwan telah mengembangkan alur kerja WGS klinis yang dapat mendeteksi peristiwa transmisi patogen sebelum menjadi dominan. Oleh karena itu, metode ini dapat secara efektif mencegah dan mengendalikan infeksi serta membantu mengembangkan strategi untuk menanggapi wabah dengan tepat.
Tentang penelitian
MRSA, VRE, ESBL-E, carbapenem-resistant Acinetobacter baumannii (CRAB), dan carbapenemase-producing Enterobacterales (CPE) diperoleh dari kultur darah, cairan serebrospinal, tempat steril, dan spesimen skrining (mis. rumah sakit di Brisbane, Australia. Sebanyak 2.660 isolat bakteri diperoleh antara 19 April 2017 dan 1 Juli 2021 dari rumah sakit yang berpartisipasi. Patogen bakteri ini diisolasi dari 2336 pasien, di antaranya 259 pasien menyediakan banyak isolat.
Pada penelitian ini pengambilan sampel dilakukan setiap minggu, dengan rata-rata 8 sampel per minggu. Sampel ini menjadi sasaran analisis WGS. WGS membantu membangun pengetikan in silico multi-locus sequence typing (MLST). Selain itu, pembuatan profil gen resistensi dilakukan menggunakan pipa analisis genom yang dipesan lebih dahulu.
Peristiwa wabah diduga ditentukan dengan membandingkan core genome single nucleotide polymorphisms (SNPs). Data klinis yang sesuai dianalisis bersama dengan data analisis genom melalui otomatisasi yang disesuaikan. Temuan ini disusun dengan laporan khusus rumah sakit yang didistribusikan secara teratur ke tim pengendalian infeksi.
Temuan studi
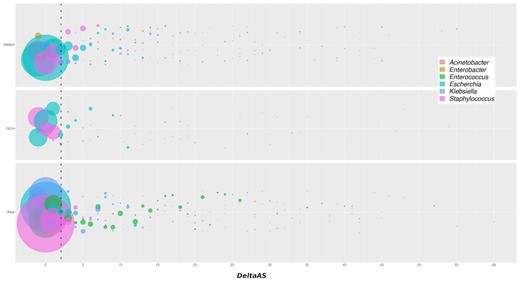
Di antara total isolat bakteri yang diurutkan selama periode penelitian, 293 ditemukan sebagai basil gram negatif MDR, 620 MRSA, dan 433 VRE. Kombinasi data genomik dan epidemiologis membantu mengidentifikasi 37 klaster yang mungkin terjadi karena komunitas, bukan peristiwa transmisi rumah sakit.
Distribution of clonal lineages among the 5 most prevalent species. Lineages with <20 representative members are classified as “other” and isolates whose sequence type had not previously been defined at the time of writing were designated as “NF.” Abbreviation: NF = Not found.
Data SNP genom inti mengungkapkan bahwa 335 isolat membentuk 76 kelompok berbeda. Menariknya, di antara 76 kluster, 43 terkait dengan rumah sakit yang berpartisipasi. Temuan ini menunjukkan terjadinya penularan bakteri yang sedang berlangsung dalam pengaturan rumah sakit. 33 klaster yang tersisa terkait dengan peristiwa penularan antar rumah sakit atau strain bakteri yang beredar dalam komunitas.
Temporal distribution and frequency of resistance-associated genotypes identified among clinical isolates. Isolates are grouped by week of collection from April 2017 to July 2021. Abbreviation: ST, sequence type.
Implikasi studi
Ketersediaan laporan yang tepat waktu sangat penting untuk mengembangkan program surveilans yang efektif. Yang penting, protokol saat ini dapat memberikan data genomik dalam waktu 10 hari setelah pengumpulan sampel. Perlu dicatat bahwa waktu penyelesaian laporan rata-rata 33 hari membatasi relevansi klinis data.
A, Epidemiological curve of all isolates collected during this study. Isolates are grouped by week of collection from April 2017 to July 2021. Dotted line indicates a change in sample collection with a focus on a targeted strategy, prioritising organisms of greatest concern (eg, CPE, CRAB, VRE, ESBL-K. pneumoniae) or invasive clinical isolates of other pathogens (eg, ESBL-E. coli, MRSA). B, Epidemiological curve of clustering isolates coloured by genus. Only the top 5 genera are shown, with all other clustering isolates classified as “Other.” Abbreviations: CPE, carbapenemase-producing Enterobacterales; CRAB, carbapenem-resistant Acinetobacter baumannii; ESBL, extended-spectrum beta-lactamase; MRSA, methicillin-resistant Staphylococcus aureus.
Beberapa faktor yang terkait dengan periode pelaporan yang lama adalah transportasi sampel yang terhambat ke laboratorium pusat, kurangnya infrastruktur WGS di tempat atau khusus, dan pengembangan pipa analisis berkelanjutan. Namun demikian, reorganisasi struktural dan penyempurnaan alur kerja dapat meminimalkan penundaan ini.
Dalam studi ini, metode berbasis WGS membantu mengidentifikasi dua cluster transmisi diduga Ab1050-A1 dan Eh90-A2, terkait dengan wabah sebelumnya. Temuan ini sangat menyarankan bahwa WGS harus dikerahkan sebagai alat surveilans prospektif untuk mencegah wabah patogen.
Community versus healthcare-association by genus. Clustering isolates are coloured by genus, as per the legend, and scaled by number of isolates. Dashed line indicates a sample collection time of 2 d post admission (DeltaAS = 2). Abbreviations: PAH, Princess Alexandra Hospital; QCH, Queensland Children's Hospital; RBWH, Royal Brisbane & Women's Hospital.
Kesimpulan
Salah satu batasan utama dari penelitian ini adalah bahwa program surveilans prospektif terutama didasarkan pada bakteri yang resistan terhadap berbagai obat. Oleh karena itu, penelitian ini gagal untuk mempertimbangkan organisme penyebab penyakit rentan antibiotik lainnya.
Meskipun sulit untuk menggabungkan alur kerja WGS dan infrastruktur komputasi lain yang sesuai dalam sistem yang ada dalam pengaturan layanan kesehatan, penting untuk menetapkan hal yang sama untuk mencegah wabah di masa mendatang. Pendirian berbasis WGD dapat mengurangi biaya keseluruhan sistem perawatan kesehatan.
Journal reference:
Forde, B. et al. (2022) "Clinical Implementation of Routine Whole-genome Sequencing for Hospital Infection Control of Multi-drug Resistant Pathogens", Clinical Infectious Diseases. doi: 10.1093/cid/ciac726. https://academic.oup.com/cid/advance-article/doi/10.1093/cid/ciac726/6691363?searchresult=1&login=false#google_vignette








No comments